▼ 価格・納期 ▼ 特徴 ▼ サンプル条件 ▼ サービス内容 ▼ 納品物 ▼ お見積もり・資料請求
Information

NEW RNA-seq年度末キャンペーン開催中!
キャンペーン期間: 2025年11月13日(木) ~ 2026年2月13日(金)ご注文分まで
キャンペーン内容: トランスクリプトーム解析(RNA-seq)を期間限定で特別価格にてご提供!
期間限定価格例: マウス 6 サンプル、遺伝子発現量比較解析込み➡¥288,000 (税別)
- ※ PolyA精製込み、取得データ量 4 Gb/サンプル(約 26 Mreads/サンプル)の場合です
- ※ 発現量比較解析ご指定例:
(#1, #2, #3) vs (#4, #5, #6)
(#1, #2) vs (#3, #4) vs (#5, #6)
#1 vs #2、#3 vs #4、#5 vs #6 など、解析パターンの数に制限はありません
- ヒト・ラット・その他生物種も対応可能です。詳細はお問い合わせください
- 作業内容:サンプルQC、ライブラリ調製、NovaSeq PE150 によるシーケンス
- データ解析内容:参照mRNA配列へのマッピング、遺伝子発現量比較解析
- 納品費用:キャンペーン期間中は無料
- 納品物:FASTQファイル、発現量一覧(Excelで閲覧可能な形式)、解析レポート(PDF)など
- 注意事項など
- → サンプル数は 6サンプルから承ります
- → 取得データ量は1サンプルあたり 4 Gb~、Gb単位でご指定可能
- → 別途ご相談ください: rRNA枯渇処理オプション、6サンプル未満でのご依頼、データ解析なしでのご依頼
お見積もり、ご相談はこちらから
ぜひお気軽にご連絡ください!
価格・納期
mRNA-Seq遺伝子発現量解析
- サンプルQC費用、Strand specificライブラリ調製費用を含みます。
- NovaSeq 2x150bp、0.13億read~/sample、2Gb~/sample
- 納期:サンプルQC合格後4~7週
※ バイオインフォマティクス解析ありの場合、+1~2週
- 参考価格:¥33,000/サンプル
マッピング+遺伝子発現量解析:+¥22,000/サンプル(参考価格)
- 出荷費用:USBメモリーの場合 ¥5,000(参考価格)、HDDの場合 ¥10,000(参考価格)
※ NovaSeq、1レーンの解析も承ります。詳しくはご相談ください。
【オプション】
| 項目 |
参考価格 |
細菌、ヒト、マウス、ラット
mRNA-Seqの為の「rRNA枯渇サービス」 |
+¥35,000 /サンプル |
| サンプルQC(2回目以降) |
+¥3,000 /サンプル |
特徴
mRNA-Seq 遺伝子発現量解析
【mRNA-Seq遺伝子発現量 × NovaSeq バリュープライス】
遺伝子配列リソースが充実した生物種における、mRNA-Seqによるサンプル間の遺伝子発現量 (Differential Expression)解析を行います。バイオインフォマティックスでは、マッピング、遺伝子発現量解析をオプション解析することができます。
- mRNA精製:ポリA精製 or rRNA枯渇
- Strand specific ライブラリ調製
- バイオインフォマティクス解析:マッピング、遺伝子発現量解析
サンプル条件
RNAサンプルのご準備に際しての注意事項は以下の通りです。
- 高品質のtotal RNAを2μg以上(濃度は20ng/μl以上)ご調製ください。
※rRNA枯渇を行う場合は、total RNAは推奨量5μg以上、必要量2.5μg以上(濃度は20ng/μl以上)ご調製ください。
- 核酸定量は、蛍光測定法をお勧めします。サンプル量が規定量に満たない場合はご相談ください。
- RNAサンプルはRNase-free水もしくは、市販RNA抽出キット付属の溶出液に溶解してください。
- 電気泳動にてサンプルに分解がないことをご確認ください。
※品質が良くない場合は、特別な処理が必要になり、追加料金を頂戴することもございます。
- DNAのコンタミネーションがないことを確認してください(DNase処理を推奨)。
- ドライアイス梱包にてクール便で送付してください。
※ドライアイスの重みでチューブが破損することを防ぐため、塊状のドライアイスは細かくして頂き、輸送時の衝撃を和らげるため、梱包材を入れて頂くことをお勧めいたします。
サービス内容
- 受領したRNAサンプルについて、品質確認(サンプルQC)を行います。
- 目的に応じたRNAの精製を行います。
ポリA精製 (*1) もしくは、rRNA枯渇処理 (*2)
- 精製したRNAの断片化を行います。
- ランダムプライマーを用いた逆転写反応により、cDNAを合成します。
- アダプター付加、2nd Strand cDNAの断片化を行い、Strand-specific mRNAライブラリーを調製します。
- 調製したライブラリーをシーケンシングします。
- (オプション解析)取得した配列を参照配列(*3)にマッピングし、遺伝子発現量の定量解析などを行います。
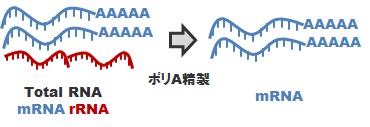
*1 mRNA-Seqライブラリ調製: ポリA精製
真核生物を対象とした標準ライブラリ調製方法です。total RNA をサンプルとしてお預かりして、ポリA精製を行い、mRNAを精製します。この mRNA を cDNA 化し、所定のアダプタを付加することにより、mRNA-Seq (Strand Specific)ライブラリを調製します。
*2 mRNA-Seqライブラリ: 細菌の場合
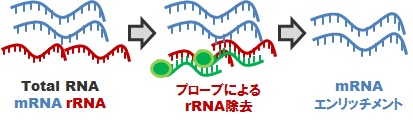
rRNA 枯渇処理 (通常はIllumina RiboZeroを使用)
原核生物(細菌)の場合、mRNA は原則として ポリA構造を持たないため、これを用いた mRNA 精製が困難です。かといって精製を行わないでシーケンシングをしても、rRNA配列ばかりを読んでしまうことになります。細菌トランスクリプトーム解析を実現するためには、予めプローブによる rRNA 枯渇処理を行ってから、cDNA 化、ライブラリ調製を行う必要があります。生物種により相性がありますので、予めご相談ください。なお、実際の rRNA 枯渇の程度については、当社で保証することはできません。
ヒト・マウス・ラットの total RNA サンプルからも、rRNAを効率よく除去することができます。断片化が進んだFFPEサンプルのrRNA除去に有効なだけではなく、ポリA精製によって失われていた、ポリA末端を持たないトランスクリプトをシーケンス解析対象とすることが可能となります。詳細については、お問い合わせください。
*3:参照配列
標準サービスでは、mRNA配列を参照配列としたマッピングを行います。
オプション解析として、参照ゲノム配列を対象としたマッピングを行います。
納品物
mRNA-Seq データのマッピング&遺伝子発現量解析
解析なしの場合
- Rawデータ(FASTQ形式)
解析ありの場合
- シーケンシング配列データ fastQ形式
- マッピング結果データ bam形式
- 遺伝子発現量データ Excelで閲覧可能な形式
※対応細菌種については、予めお問い合わせください。
お見積りはコチラ
▲ ページ先頭へ戻る