▼ サービス内容 ▼ サンプルのご準備方法 ▼ 参考料金 ▼ 関連資料
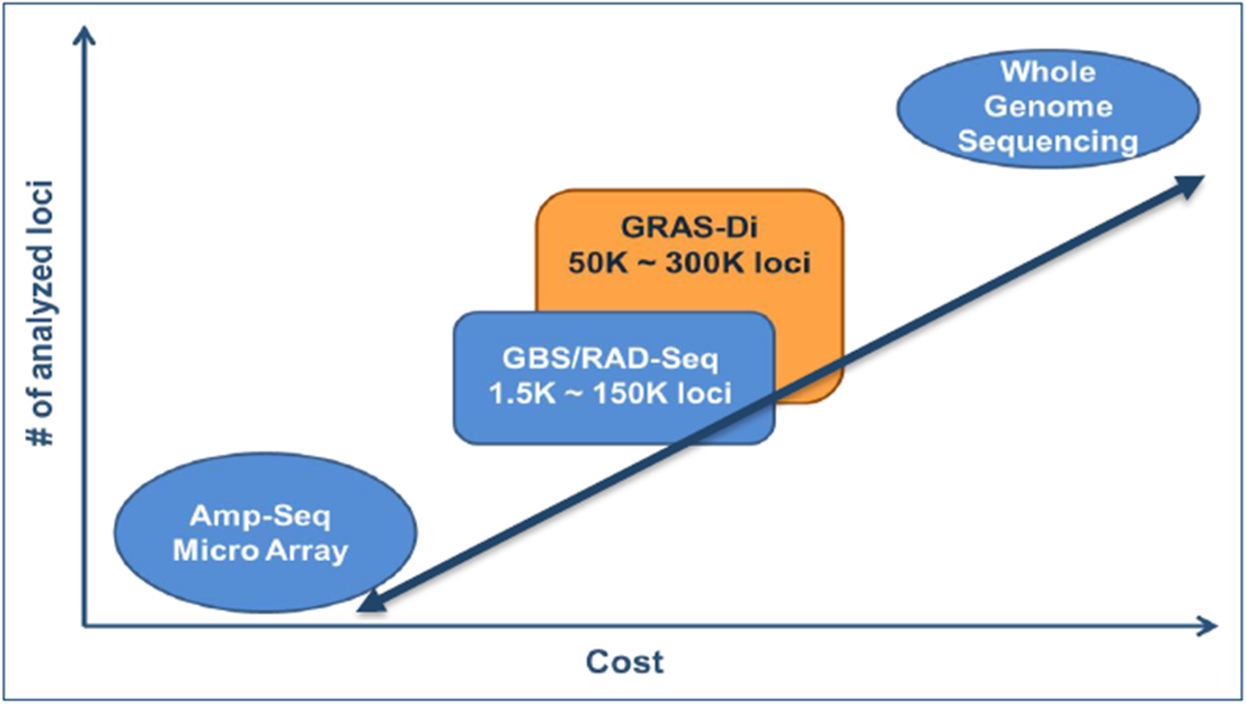
ゲノムワイドなジェノタイピング
ゲノム育種や自然集団解析をサポート。
5つの特徴
GBSやRAD-Seqに比べて解析領域の偏りが少なく、
ゲノム上一様なジェノタイピングが可能。
アンプリコン増幅の再現性が非常に高く、
ジェノタイプデータの欠陥が少ない。
ゲノム情報が利用可能な生物種では、
アンプリコンマーカーのマッピングとSNP抽出も可能。
リファレンス配列が無い生物種においても ジェノタイピングが可能。
分離集団または自然集団においても ジェノタイピングが可能
こんなお悩みにぴったり!
- 大規模にマーカー探索を行いたいけど、サンプル数も多く全てゲノム解析するほど予算が十分でない
- これまでもGBSやRAD-Seqを試してみたが、期待するほどマーカー数が得られなかった
- 将来的にはターゲットを絞ってアンプリコンシーケンスを行いたいので、まずは候補マーカーを数多く得ておきたい
お問い合わせはコチラ
サービス内容
豊富な解析実績、国内外から120生物種以上、70,000サンプル以上(2022年9月時点)

- 【内容】ライブラリ調製からシーケンス、データ解析まで
GRAS-Di®ライブラリ調製
NovaSeqによるシーケンス
期待リード数:平均 5 Mread/sample
GRAS-Di®解析ソフトによるシーケンスデータの解析(アンプリコンの有無をベース
にしたジェノタイピング結果、アンプリコンマーカーのマッピング結果)
- 【オプション】
ゲノム情報が利用可能な生物種では、バイオインフォマティクスのご相談を承ります。
(SNP抽出)
※解析可能な生物種についてはお問合せください。
- 【納品物】
シーケンシング配列データ(fastq形式)
GRAS-Di®解析ソフトによる解析結果(Excelで閲覧可能な形式)
※GRAS-Di紹介動画も併せてご参照ください
共優性マーカーの詳細情報 ※
アンプリコンのマッピング位置情報 ※
※解析のご対応可能な場合に納品させて頂きます。
サンプルのご準備方法
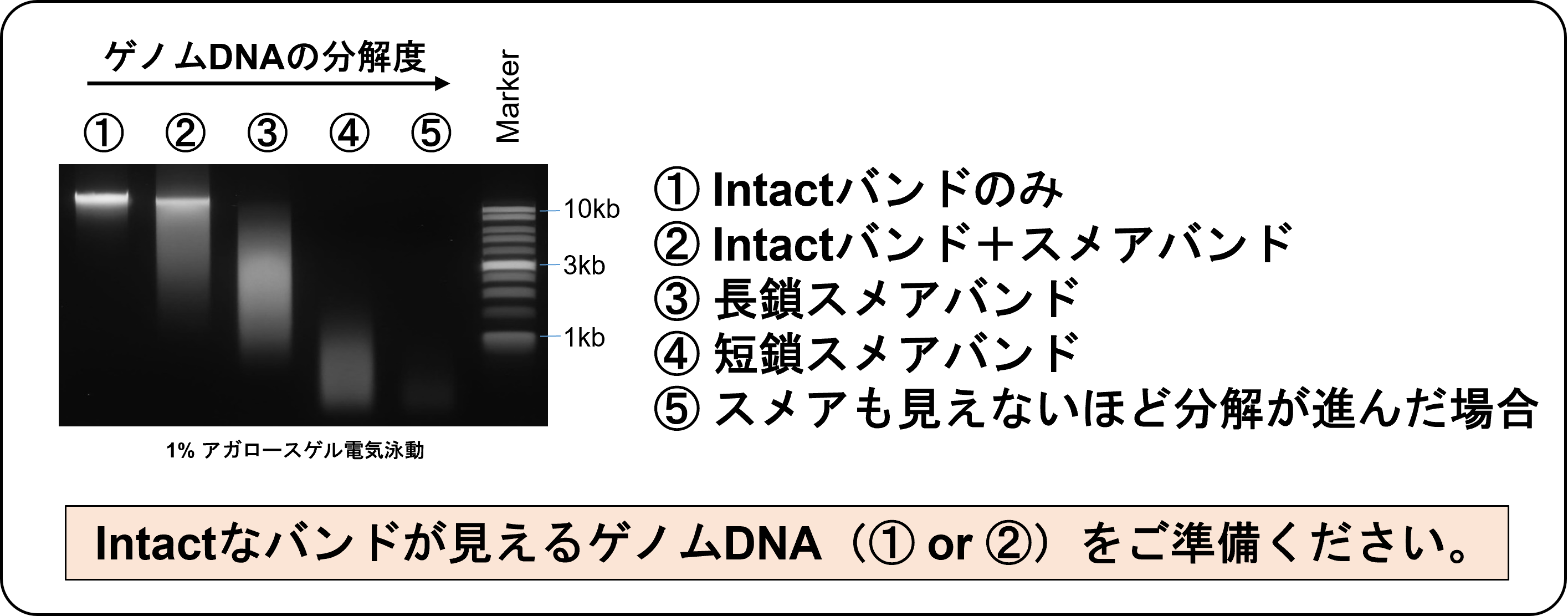
- ●TEへ溶出したgDNA ( >30ng/ul, >20ul)をご準備いただきお送りください。
●濃度はUVを用いた吸光光度法により測定・ご確認ください。
(蛍光測定法による定量はご遠慮ください)
●冷蔵便もしくは冷凍便にてサンプルDNAはご送付ください。
※ サンプルが規定量に達しない場合はご相談ください。
※ 電気泳動法などを利用し、RNAのコンタミネーションがないこと、gDNAが分解
していないことをご確認ください。
-
サンプル発送時の注意事項
指定のチューブあるいはプレートでお送り下さい。ご用意が難しい場合は発送前にご連絡下さい。
48サンプル以上の場合は、取り扱い時のミスを避ける為にプレートでお送りください。
プレートの場合、8連キャップにて密閉し、液漏れがないことを確認の上、ご発送ください。
追加作業の必要性が生じた場合は、納期の変更、あるいは追加料金が発生することもあること、予めご了承下さい。
参考料金
ご計画の対象生物、サンプル数をお知らせください。最適なプランをご提案いたします。
| 仕様例※ |
参考価格 |
1サンプル相当 |
納期 |
48サンプル
250Mreads/total、データ解析あり |
¥330,000 |
約6,800円 |
サンプルQC合格後
7〜10週間 |
96サンプル
500Mreads/total、データ解析あり |
¥490,000 |
約5,100円 |
サンプルQC合格後
7〜10週間 |
96サンプル
500Mreads/total、データ解析なし |
¥440,000 |
約4,600円 |
サンプルQC合格後
6〜9週間 |
1000サンプル
NovaSeq6000 1レーン、データ解析あり |
ご相談 |
ご相談 |
サンプルQC合格後
9~12週間 |
1000サンプル
NovaSeq6000 1レーン、データ解析なし |
ご相談 |
ご相談 |
サンプルQC合格後
9~12週間 |
※上記はいずれも96ウェルプレートでサンプルをご提出頂く場合の価格・納期です。
シングルチューブでサンプルをご提出頂く場合の価格・納期はご相談ください。
(参考)シングルチューブご利用の場合:追加料金+500円(参考価格)/サンプル、追加納期:+2週間
● トヨタ自動車社開発 GRAS-Di解析ソフトウェア
※インストーラー、ユーザーズマニュアル(印刷物)を納品いたします。
関連資料
関連ページ
その他資料
-
GRAS-Di®をご利用いただいた論文
- 参考文献ページから各論文へ移行できます
(1)弊社サービスをご利用いただいた論文
- Genomic evidence for speciation with gene flow in broadcast spawning marine invertebrates.
Hirase S, Yamasaki YY, Sekino M, Nishisako M, Ikeda M, Hara M, Merilä J, Kikuchi K. (2021)
Mol Biol Evol. doi: 10.1093/molbev/msab194.
- Effect of Three Types of Ion Beam Irradiation on Gerbera (Gerbera hybrida) In Vitro Shoots with Mutagenesis Efficiency.
Hosoguchi T, Uchiyama Y, Komazawa H, Yahata M, Shimokawa T, Tominaga A. (2021)
Plants. 10:1480.
- Major and Stable Quantitative Trait Locus qSS2 for Seed Size and Shape Traits in a Soybean RIL Population.
Kumawat G, Xu D. A. (2021)
Front Genet. 12:646102.
- QTL analysis of crown gall disease resistance in apple: first plant R gene candidates effective against Rhizobium rhizogenes (Ti).
Moriya, S., Iwanami, H., Haji, T. et al. (2021)
Tree Genetics & Genomes 17:25.
- Genetic Dissection of a Precocious Phenotype in Male Tiger Pufferfish (Takifugu rubripes) using Genotyping by Random Amplicon Sequencing, Direct (GRAS-Di).
Yoshikawa S, Hamasaki M, Kadomura K, Yamada T, Chuda H, Kikuchi K, Hosoya S. (2021)
Mar Biotechnol (NY). doi: 10.1007/s10126-020-10013-4.
- Random PCR-based genotyping by sequencing technology GRAS-Di (genotyping by random amplicon sequencing, direct) reveals genetic structure of mangrove fishes.
Hosoya S, Hirase S, Kikuchi K, Nanjo K, Nakamura Y, Kohno H, Sano M.
Mol Ecol Resour. 2019 Sep; 19:1153-1163. doi: 10.1111/1755-0998.13025.
(2)GRAS-Di技術ご利用論文(弊社以外でのサービスご利用者様を含む)

-
対応生物種 豊富な解析実績 国内外から120生物種以上、70,000サンプル以上 (2022年9月時点)
- トヨタ自動車株式会社様 検証済み対応生物種(58生物種)
イネ
コムギ
トウモロコシ
サトウキビ
ダイズ
ラッカセイ
エンドウ
トマト
ナス
ピーマン
パプリカ
ジャガイモ
サツマイモ
キャベツ
レタス
ハクサイ |
ダイコン
ブロッコリー
カリフラワー
ネギ
タマネギ
キュウリ
カボチャ
ホウレンソウ
ニンジン
ゴボウ
イチゴ
メロン
スイカ
サブクローバー
ホースグラム |
ミヤコグサ
リンゴ
モモ
ナシ
オレンジ
カキ
ブドウ
イチジク
キウイ
ユズ
スダチ
プルーン
キク
スギ
サクラ |
ヒト
ウシ
ブタ
ニワトリ
ウマ
ウサギ
ハムスター
モルモット
ラット
マグロ
シイタケ
マイタケ |
ユーロフィンジェノミクス株式会社 受託実績 (数万検体以上)※
植物 (イネ、かんきつ類、バラ類、アブラナ科、セリ科)
動物 (魚類、貝類、昆虫類) |
※一度に1,000検体以上のご依頼も承ります。
お問い合わせはコチラ
- GRAS-Di®技術はトヨタ自動車株式会社にて開発されました。
- GRAS-Di®はトヨタ自動車株式会社の登録商標です。
- GRAS-Di(グラス ディーアイ) Genotyping by Random Amplicon Sequencing-Direct
▲ ページ先頭へ戻る